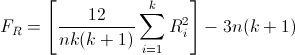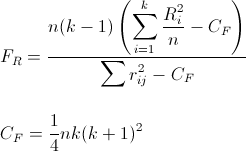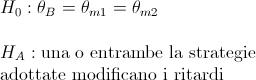Il test Q di Cochran rappresenta una estensione del test di McNemar, il quale può essere applicato con analoghi risultati quando k = 2.
Ma vediamo subito un esempio, per comprendere il campo di applicazione di questo test.
Una società svolge una ricerca di mercato, e chiede a 12 soggetti di compilare un questionario. Tra le domande, figura il quesito: "acquisteresti il prodotto X da una o più delle seguenti 3 marche (A, B, C)?". I 12 soggetti rispondono con delle crocette per marcare la loro preferenza di marca. Le possibili risposte variano da "nessuna crocetta" a "tre crocette" sulle 3 marche.
Risulta subito evidente che i dati di cui disponiamo sono binomiali (categorici): 0 indica che la marca in questione non è stata barrata, mentre 1 indica che la marca è stata crocettata.
La tabella seguente presenta i dati che vengono così raccolti:
Le ipotesi del problema sono: la proporzione di "sì" per ciascuna condizione sono uguali; l'ipotesi alternativa è che almeno una delle proporzioni differisce dalle altre.

La statistica test è:

I simboli che qui compaiono sono:
k = numero di gruppi (o condizioni);

La tabella seguente mostra i calcoli che vengono eseguiti:
Calcoliamo quindi il Q di Cochran:

Il Q di Cochran segue una distribuzione Chi-quadro, con k-1 gradi di libertà. Il valore critico tabulato è:
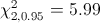
Poichè Q è maggiore di Chi-quadro, rifiutiamo l'ipotesi nulla H0.
Si pone adesso il problema di identificare quale/i gruppo/i differisce significativamente dagli altri, rendendo quindi significativo il test Q.
Per fare questo, possiamo eseguire il test di McNemar, a coppie. Del test ne parlerò in un prossimo post, ma è sufficiente una rapida ricerca su Google per ottenere pagine esaustive.
Calcolando il test di McNemar per le 3 coppie di paragone, dobbiamo correggere il livelli di significatività (in quanto stiamo eseguendo pur sempre dei confronti multipli). Poichè le coppie da analizzare sono 3, dividiamo il valore di alpha per 3, e quindi il livello di significatività deve essere fissato pari a 0.0167.
Soluzione in R
mydata <- matrix(c(
1, 1, 0,
0, 1, 0,
1, 1, 1,
0, 1, 0,
0, 1, 0,
0, 1, 1,
0, 0, 0,
0, 1, 0,
1, 1, 0,
0, 1, 0,
0, 0, 0,
0, 0, 1),
nrow = 12,
byrow = T,
dimnames = list(1 : 12,
c("MarcaA", "MarcaB", "MarcaC")))
Utilizziamo il seguente codice, che ho trovato a questo link:
cochranq.test <- function(mat)
{
k <- ncol(mat)
C <- sum(colSums(mat) ^ 2)
R <- sum(rowSums(mat) ^ 2)
T <- sum(rowSums(mat))
num <- (k - 1) * ((k * C) - (T ^ 2))
den <- (k * T) - R
Q <- num / den
df <- k - 1
names(df) <- "df"
names(Q) <- "Cochran's Q"
p.val <- pchisq(Q, df, lower = FALSE)
QVAL <- list(statistic = Q, parameter = df, p.value = p.val,
method = "Cochran's Q Test for Dependent Samples",
data.name = deparse(substitute(mat)))
class(QVAL) <- "htest"
return(QVAL)
}
Applichiamo la funzione:
cochranq.test(mydata)
Cochran's Q Test for Dependent Samples
data: mydata
Cochran's Q = 8, df = 2, p-value = 0.01832
In alternativa, possiamo usare la funzione cochran.test della library outliers.
Adesso analizziamo le coppie di gruppi con il McNemar's Chi-squared Test for Count Data:
mydatadf <- as.data.frame(mydata)
AB <- with(mydatadf, table(MarcaA, MarcaB))
BC <- with(mydatadf, table(MarcaB, MarcaC))
AC <- with(mydatadf, table(MarcaA, MarcaC))
mcnemar.test(AB)
McNemar's Chi-squared test with continuity correction
data: AB
McNemar's chi-squared = 4.1667, df = 1, p-value = 0.04123
mcnemar.test(BC)
McNemar's Chi-squared test with continuity correction
data: BC
McNemar's chi-squared = 3.125, df = 1, p-value = 0.0771
mcnemar.test(AC, correct=T)
McNemar's Chi-squared test
data: AC
McNemar's chi-squared = 0, df = 1, p-value = 1